

Antropogenética
Práctica O3. Tratamientos de datos II (La
población subdividida).
Base de datos de MAPT con los resultados de las prácticas:
DBFrecuencias-MAPT.xls
En esta práctica se tendrán en cuenta las frecuencias de 5 genes HLA
analizados con alta resolución en varios cantones de Suiza.
El trabajo de referencia es:
Buhler
S, Nunes JM, Nicoloso G, Tiercy JM, Sanchez-Mazas A. (2012) The
heterogeneous HLA genetic makeup of the Swiss population. PLoS One.
7(7):e41400.
Los genes son HLA-A, -B, -C, -DRB1 y -DQB1.
La base de datos con las frecuencias alélicas se encuentran en la
sección Suppoting Information
del propio trabajo.
Los programas que van a utilizarse son:
GeDis
- Manual
del programa (pdf)
Past
Arlequin 3.5 - Programa y manual
Arlequin
3.11
GeDis trae incluída como ejemplo precisamente esta base de datos, de
modo que no es preciso copiarla del original.
Descargue el programa en su carpeta de prácticas.
La situación de las poblaciones representativas de los cantones analizados se muestra en el mapa, así como la distribución de los idiomas hablados en todos ellos, incuyendo alemán, francés, italiano y romanche. Suponen el 63,7%, 20,4%, 6,5% y 0,5% de la población, respectivamente.

En la carpeta GeDis se encuentran las frecuencias alélicas en la primera hoja del fichero Data.xls.
El formato de la primera hoja del fichero Data.xls es el
siguiente:
Casilla A1: Número de poblaciones.
Casilla B1: Número de marcadores genéticos utilizados.
Fila 2, desde la casilla A2 en adelante: Número de alelos de cada
marcador, hasta el total de marcadores especificados en la casilla B1.
Fila 3, desde la casilla B3 en adelante: Etiquetas de las poblaciones.
No se utiliza la casilla A3.
Fila 4, en la casilla A4, etiqueta del primer alelo y desde la casilla
B4 en adelante, frecuencias del alelo en todas las poblaciones.
Fila 5 y sucesivas, hasta el número total de alelos indicados en la
fila 2, igual formato que la fila 4.
Arranque el programa GeDis pulsando dos veces sobre el icono.
Lea el
fichero de datos (Data.xls) mediante la opción "Input" y "Read
data.xls". Compruebe que ha leído bien los datos, de modo que no falta
ninguna población o ningún alelo y que no ha leído todos los valores
como 0, por incompatibilidad entre puntos y comas.
Obtenga la matriz de distancias Fst de Reynolds mediante la opción
"Distances" y "MDS-Reynolds'Fst". Compruebe que es una matriz de
distancias, simétrica y con valores 0 en la diagonal. No la cierre.
Realice un Análisis de Escalamiento Multidimesional mediante la opción
"MDS"
y "MDS-Reynolds'Fst"
El gráfico MDS aparecerá guardado en la carpeta outputs como
MDSFst.eps, que puede insertar en su fichero Word de resultados.
Interprete el gráfico de Escalamiento Multidimensional a partir de los
datos que conoce.
Abra de nuevo la hoja de cálculo Data.xls y observe la hoja 2, que
tiene las coordenadas geográficas de las poblaciones con el siguiente
formato:
Casilla A1: Número de poblaciones, como en la primera hoja.
Fila 2, desde la casilla A2 en adelante: Etiquetas de las poblaciones.
Deberán ser las mismas etiquetas de la hoja 1 y en el mismo orden.
Fila 3, desde la casilla A3 en adelante: Grados de latitud para cada
población.
Fila 4, desde la casilla A4 en adelante: Minutos de latitud para cada
población.
Fila 5, desde la casilla A5 en adelante: 1 para latitud Norte y -1 para
latitud Sur.
Fila 6, desde la casilla A6 en adelante: Grados de longitud para cada
población.
Fila 7, desde la casilla A7 en adelante: Minutos de longitud para cada
población.
Fila 8, desde la casilla A8 en adelante: 1 para longitud Este y -1 para
longitud Oeste.
Para buscar las coordenadas geográficas de las poblaciones analizadas
con GeDis se puede utilizar Google Maps con la opción "¿Qué hay aquí?"
Mediante el programa GeDis obtenga, pulsando en las diferentes opciones
del menú "GeoGraphics":
- Un listado de clinas con su orientación y su significación. El
fichero con la imagen de las clinas se llama Clines.eps.
- Un análisis de Autocorrelación Espacial. El fichero con el gráfico de
la autocorrelación se llama Autocorrelation.eps.
- Un análisis del flujo génico mediante el método del centroide. El
fichero con el gráfico correspondiente se llama Centroid.eps.
- Un gráfico con la regresión de la clina correspondiente al alelo A*02.
- Un mapa sintético para este mismo alelo.
- La clina y el mapa sintético del alelo DQB1*3:1/9/19/21.
- Un mapa sintético para Ri.
- Un mapa sintético para los 3 primeros vectores propios del MDS-Fst.
Salve los resultados con la opción "Outputs" y "Save Results".
Aparecerán en el fichero Results.txt.
Interprete los resultados obtenidos.
Posición geográfica. Test de Mantel entre
matrices geográficas y genéticas.
Ejecute GeDis y obtenga la matriz de distancias geográficas:
"Input" y "Read Data.xls"
"Distances" y "Geographical"
Copie la matriz en Past, seleccionando primero "Edit labels" y
compruebe que la configuración es correcta. Para ello, basta con
seleccionar toda la matriz, elegir "Multivar y "Non metric MDS" y
especificar "User distance". Aparecerá una configuración similar al
mapa, aunque puede estar girada o invertida.
Obtenga ahora la matriz de distancias Fst.
"Distances" y "Reynolds'Fst"
Copie la matriz en Past debajo de la matriz de distancias geográficas.
Seleccione la primera matriz y adjudiquele un color ("Edit" y "Row
color/symbol"). Después seleccione la otra matriz y adjudiquele otro
color.
Seleccione todos los datos (pulse en el nombre de la primera población
y después, pulsando mayúsculas, pulse en el nombre de la última) y
realize un test de Mantel de comparación entre ambas matrices
("Multivar" y "Mantel test"). Especifique "User distance" en ambas
matrices y "Calculate".
Interprete el resultado. Compárelo con los análisis del aislamiento por
la distancia y la autocorrelación espacial.
Barreras geográficas. AMOVA entre Alpes y
Meseta.
Abra el fichero de resultados de la práctica anterior. Compare el
resultado del MDS con la distribución geográfica de los cantones entre
la región alpina, en relación a la meseta central y el Jura, en los
siguientes mapas:
 |
 |
|---|
Ejecute en GeDis la opción "Output" y "Export to Arlequin".
Se
obtienen 5 ficheros, llamados infile1.arp hasta infile5.arp,
correspondientes a cada uno de los genes. Son los ficheros con los
datos de las frecuencias en cada población, con el formato específico
de Arlequin.
Añada al final de los 5 ficheros el siguiente texto, abriéndolos desde
Word:
[[Structure]]
StructureName="Cantones de
Suiza: Alps vs Plateau & Jura "
NbGroups=2
#Plateau
Group={
"Aarau"
"Bern"
"Basel"
"Luzern"
"St-Gallen"
"Zurich"
"Geneve"
"Lausanne"
}
#Alpes
Group={
"Sion"
"Lugano"
"Coira"
}
No es preciso que cierre los ficheros. Basta con salvarlos,
manteniendo el formato "sólo texto".
Arranque Arlequin. Abra sucesivamente los 5 ficheros ("Open Project"),
comprobando que la estructura de grupos es correcta. Especifique en
"Settings" la opción AMOVA ("Standard AMOVA computations") y ejecute
las instrucciones con "Start".
Hemos especificado 2 grupos poblacionales, correspondientes a los Alpes
y a la Meseta/Jura y hemos realizado un Análisis Molecular de la
Varianza para cada uno de los 5 genes.
Los resultados aparecerán en 5 carpetas diferentes con los nombres
infile1.res a infile5.res. Se pueden revisar abriendo el fichero
infile1_main.htm con el navegador. Cierre después estos ficheros.
Haga una tabla de AMOVAs e interprete los resultados.
Barreras lingüísticas. AMOVA entre
germanófonos, francófonos, italianófonos y romances.
Otro factor interesante para la interpretación de los resultados puede
ser el lingüístico.
Abra el fichero de resultados de la práctica anterior. Compare el
resultado del MDS con la distribución de los cantones con diferentes
idiomas en el siguiente mapa:
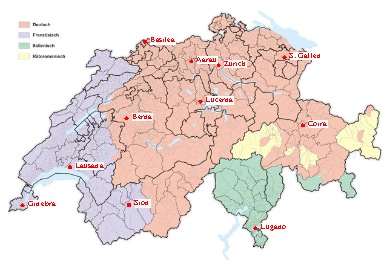
Sustituya la estructura de grupos de los 5 ficheros infile por la siguiente y sálvelos:
[[Structure]]
StructureName="Cantones de
Suiza: Idiomas"
NbGroups=4
#Aleman
Group={
"Aarau"
"Bern"
"Basel"
"Luzern"
"St-Gallen"
"Zurich"
}
#Frances
Group={
"Geneve"
"Lausanne"
"Sion"
}
#Italiano
Group={
"Lugano"
}
#Romance
Group={
"Coira"
}
Abra sucesivamente los 5 ficheros ("Open Project"),
comprobando que
la estructura de grupos es correcta. Especifique en "Settings" la
opción AMOVA ("Standard AMOVA computations") y ejecute las
instrucciones con "Start".
Hemos especificado 4 grupos
poblacionales, correspondientes a los 4 idiomas y hemos realizado un
Análisis Molecular de la Varianza para cada uno de los 5 genes.
Abra de nuevo los ficheros infile1_main.htm con el navegador.
Haga una tabla de AMOVAs e interprete los resultados.
Tamaño de población.
Teniendo en cuenta que
los tamaños de población de los cantones representados por las 11
ciudades en el análisis son notablemente diferentes, como se observa en
la siguiente tabla,
|
Ciudad
|
Habitantes
|
|---|---|
|
Aarau
|
819285
|
|
Bern
|
969299
|
|
Basel
|
451400
|
|
Geneve
|
441000
|
|
Coira
|
187803
|
|
Lugano
|
319800
|
|
Lausanne
|
657700
|
|
Luzern
|
381966
|
|
St-Gallen
|
459999
|
|
Sion
|
288800
|
|
Zurich
|
1273278
|
Interprete la posición en el MDS y en el análisis del centroide de Zurich por un lado y Sion, Lugano y Coira por otro. Puede resultar útil representar en un gráfico de dispersión el tamaño poblacional respecto a los valores de Ri, que se encuentran en el fichero Results.txt de Gedis.
Envíe los resultados en un correo@Jose A. Peña, Mikel Iriondo, 2007-2024 Universidad del País Vasco