-

La UPV/EHU celebra el acto de investidura de sus 512 nuevos doctores y nuevas doctoras de 2023
-

El lingüista Joaquín Gorrochategui, Premio Euskadi de Investigación 2023
-

Francisco Etxeberria recibe el I Premio Conchita Viera
-

El ejercicio físico supervisado mejora el bienestar de los cuidadores de personas mayores
-

El poder de las metáforas
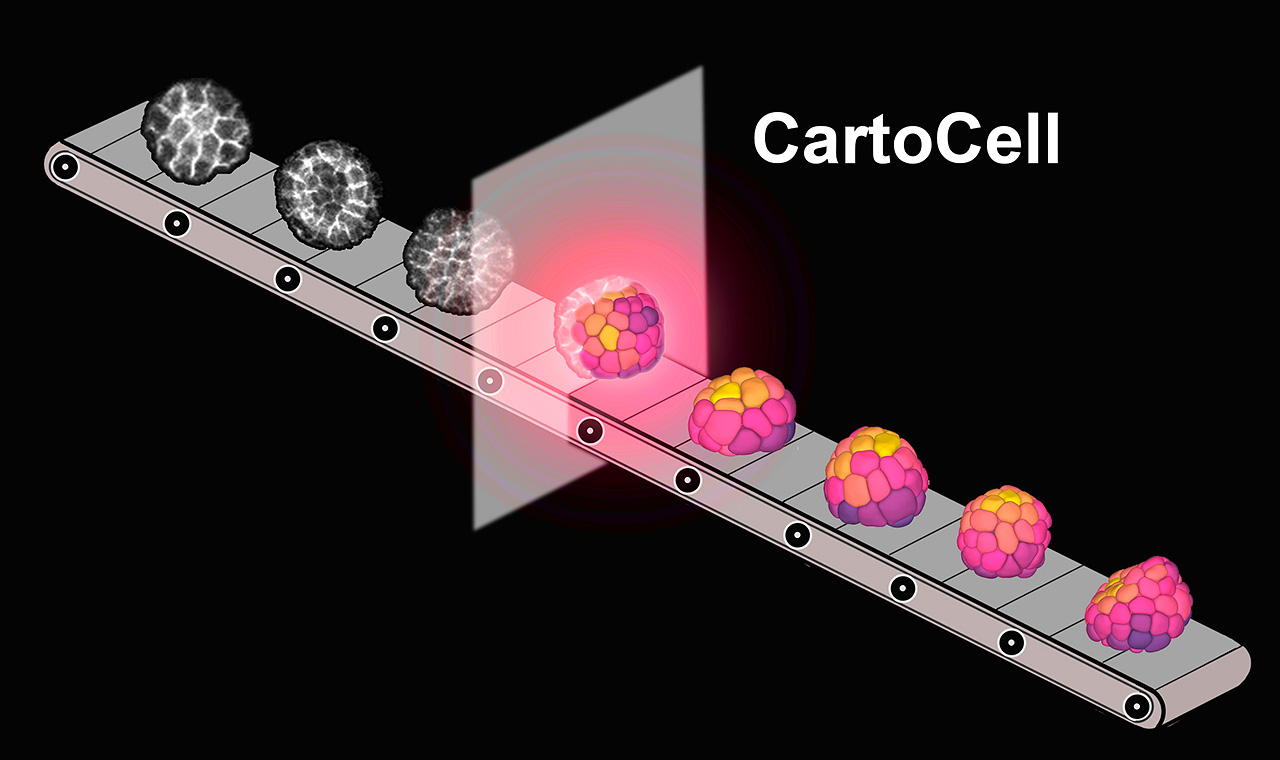
Desarrollan una herramienta de código abierto capaz de detectar patrones ocultos en los tejidos celulares
En el proyecto CartoCell, que analiza imágenes en 3D con inteligencia artificial, ha colaborado la Universidad del País Vasco, el Instituto Biofisika y el DIPC
- Investigación
Fecha de primera publicación: 25/09/2023

Una reciente colaboración entre el Instituto de Biomedicina de Sevilla (Hospital Universitario Virgen del Rocío/CSIC/Universidad de Sevilla) con la Universidad del País Vasco, el Instituto Biofisika, el Donostia International Physics Center y otros prestigiosos centros nacionales e internacionales, han dado como resultado la publicación en la revista Cell Reports Methods de CartoCell, una herramienta de análisis de imágenes en 3D de alto contenido que revela patrones de morfología celular escondidos. Este software, de código abierto, tiene un gran potencial de aplicación tanto en ciencia básica, con la que ya ha sido probado, como para el desarrollo clínico y médico.
En busca de patrones ocultos
Los animales y plantas, además de otras estructuras de la naturaleza, presentan patrones que podemos detectar fácilmente: las rayas de las cebras, los polígonos de la jirafa o en las conchas de las tortugas, por ejemplo. Todos estos patrones tienen en común que son macroscópicos. Sin embargo, este mismo fenómeno se da a nivel microscópico, en los tejidos. “Estos patrones también aparecen durante el desarrollo embrionario”, explica Luis M. Escudero, uno de los autores e investigador del Instituto de Biomedicina de Sevilla (IBiS) y profesor de la Universidad de Sevilla. “Por ejemplo, hay genes que se expresan en patrones alternos de rayas y que son fundamentales para establecer los segmentos de los insectos”. Muchos de dichos patrones son ya conocidos y muy estudiados, sin embargo, queda mucho por descubrir.
“En nuestro trabajo hemos encontrado patrones que aún estaban escondidos…. Y que solo han podido ser desvelados al usar herramientas de inteligencia artificial para el análisis de imágenes microscópicas de tejidos. Vemos que, incluso en estructuras muy simples, ya hay asimetrías en la morfología de las células. Esta es la forma en que comienza la morfogénesis”. La morfogénesis se refiere al proceso biológico que causa que un organismo desarrolle su forma y estructura. Es una de las etapas fundamentales durante el desarrollo embrionario y se refiere a la organización y distribución espacial de las células diferenciadas para formar estructuras específicas y eventualmente órganos completos.
CartoCell, una herramienta para cartografiar los tejidos epiteliales
“CartoCell es una herramienta software que permite procesar de manera rápida y automática una gran cantidad de imágenes 3D obtenidas en el microscopio para la reconstrucción y el análisis de tejidos epiteliales a nivel celular”, prosigue el Dr. Escudero. En otras palabras, esta herramienta digital es capaz de trabajar de forma automática muchísima información, especializándose en buscar patrones, formas, distribuciones y estructuras en los tejidos a partir de imágenes.
Según aclara, CartoCell utiliza la potencia de las redes neuronales, específicamente una red neuronal artificial diseñada para optimizar la identificación de células tridimensionales en imágenes de microscopía. La principal ventaja de CartoCell es que puede identificar y estudiar la forma y distribución espacial de cada célula en el tejido epitelial, permitiendo descubrir patrones asociados a diferentes tipos de tejidos.
“Cada célula del tejido es reconstruida con una gran fidelidad, gracias a lo cual podemos estudiar tanto su forma como su distribución espacial. De este modo, CartoCell nos permite descubrir patrones asociados a los diferentes tipos de tejidos y células, así como estudiar sus reglas de empaquetamiento”. De momento, el software ha sido usado para estudios de ciencia básica, permitiendo cartografiar la morfología de las células con imágenes de diferentes tipos de tejidos. “De este modo, hemos podido encontrar patrones ocultos de una manera sencilla y muy visual”, continúa el investigador, “lo que es fundamental para estudiar la organización de tejidos epiteliales donde las células presentan un contacto estrecho”.
Pero, además de los patrones ocultos, y de la investigación biológica de tejidos, CartoCell también muestra un gran potencial de aplicación clínica. “En el ámbito biomédico, la posibilidad de analizar una gran cantidad de muestras de manera rápida y fiable, como hace CartoCell, es ideal para evaluar la reproducibilidad de cultivos de organoides [pequeños tejidos creados para simular un pequeño órgano] epiteliales y realizar comparativas detalladas entre condiciones normales y patológicas. Por poner un ejemplo sencillo”, continúa el investigador, “el testeo del efecto de fármacos en tejidos animales o humanos podría beneficiarse de nuestro método. Nuestro avanzado análisis de imágenes podría detectar cambios sutiles a nivel celular, que a la larga pueden ser importantes, del efecto de cada fármaco contra una determinada enfermedad”.
Redes neuronales y código abierto en su núcleo
El corazón de CartoCell es una red neuronal artificial. La arquitectura de la red fue diseñada partiendo de otros modelos desarrollados anteriormente por el grupo de investigación en visión artificial para bioimagen del Instituto Biofisika y el Donostia International Physics Center (DIPC), en el País Vasco, que participa en el proyecto. “Estos modelos ya habían demostrado gran versatilidad y robustez para problemas similares en imagen de microscopía de diferentes modalidades (microscopía electrónica y microscopía confocal, entre otras)”, explica el doctor Ignacio Arganda, líder de dicho grupo e investigador Ikerbasque afiliado también a la Facultad de Informática de la Universidad del País Vasco (UPV/EHU). “En concreto, la arquitectura y las imágenes de salida que genera fueron adaptadas para optimizar la identificación de células tridimensionales adquiridas mediante técnicas de microscopía”.
“Como toda red neuronal artificial”, continúa, “la nuestra necesita del conocido como conjunto de entrenamiento para aprender a realizar la tarea que le propongamos. En nuestro caso, dicha tarea se trata de la identificación (o “segmentación”) de células epiteliales en imágenes tridimensionales”. Normalmente, para que una red neuronal sea capaz de generalizar y realizar esta tarea con precisión, necesita un conjunto de datos muy grande y heterogéneo. Construir dicho conjunto requiere muchísimo trabajo manual, ya que habría que “pintar” una a una todas las células de las imágenes 3D de entrenamiento.
“Sin embargo”, aclara el experto, “CartoCell utiliza un conjunto de entrenamiento inicial de epitelios pintados muy pequeño. Con ese conjunto, entrenamos nuestra red neuronal una primera vez, e intentamos que prediga las identidades celulares en muchas otras imágenes de epitelios. Como es esperado, el resultado no es el ideal, pues muchas identidades no están completas, y quedan huecos entre las células. Para solucionarlo, empleamos un algoritmo de Voronoi que rellena los espacios vacíos entre las células identificadas, consiguiendo así que contacten entre ellas".
“Estas etiquetas celulares transformadas son las que llamamos weak labels [etiquetas débiles], y las utilizamos como nuestro nuevo conjunto de entrenamiento. Aunque estas etiquetas no son totalmente perfectas, capturan de una forma bastante realista la morfología celular (dado que las células epiteliales comparten algunas características de organización con los patrones de Voronoi). Además, al ser muchas y muy heterogéneas, ayudan a mejorar el proceso de re-entrenamiento de la red. Este paso es clave en CartoCell, ya que nos permite conseguir muy buenos resultados en segmentación sin tener que emplear un tiempo excesivo en etiquetar manualmente un conjunto de entrenamiento grande”, completa el investigador.
Una de las características de CartoCell es su versatilidad, según defiende el Dr. Pedro Gómez-Gálvez, autor también del trabajo y actualmente en el Departamento de Fisiología, Desarrollo y Neurociencia de la Universidad de Cambridge (Reino Unido). En concreto, su red neuronal se podría sustituir por cualquier modelo de segmentación del estado del arte (redes neuronales convolucionales 3D o cualquiera de sus variantes), siempre y cuando se utilice el concepto de etiquetado ligero o “weak labels” anteriormente descrito para mejorar el conjunto de entrenamiento. “De hecho, en nuestro artículo probamos con varias arquitecturas de red modernas y, con todas, los resultados fueron muy buenos tras el etiquetado ligero siguiendo el algoritmo de Voronoi”, confirma Gómez-Galvez.
Además, CartoCell es una herramienta de código abierto, lo que brinda la oportunidad a otros expertos de utilizar, mejorar o adaptar libremente su uso. “Todas las personas que hemos participado en la creación de CartoCell creemos firmemente en la idea de ciencia abierta. Consecuentemente, CartoCell ha sido implementado desde el primer día como código abierto, y siempre ha estado disponible para la comunidad. En ese sentido, nuestra intención ha sido crear una herramienta accesible, fácil de instalar y utilizar por personas no expertas, pero también adaptable y ampliable para quien quiera desarrollar nuevas soluciones sobre ella”, zanja.
Además del Instituto de Biomedicina de Sevilla y de la Universidad del País Vasco, el Instituto Biofisika, el Donostia International Physics Centers, en este trabajo han participado también investigadores de la Universidad de Sevilla, el Centro de Biología Molecular Severo Ochoa de Madrid, y el MRC Laboratory of Molecular Biology de Cambridge en el Reino Unido, entre otras instituciones.
Referencia bibliográfica
- CartoCell, a high-content pipeline for 3D image analysis, unveils cell morphology patterns in epithelia
- Cell Reports Methods
- DOI: 10.1016/j.crmeth.2023.100597